冷冻电镜中心助力冯钰课题组在Nature Plants发文揭示植物microRNA加工的分子机制
2021-10-09 16:47 发布人:系统管理员冯钰课题组在Nature Plants发文揭示植物microRNA加工的分子机制
编辑:林海燕 来源:基础医学系
2021年9月30日,浙江大学基础医学院、浙江大学医学院附属邵逸夫医院冯钰课题组在Nature Plants在线发表了题为“Structural basis of microRNA processing by Dicer-like 1”的研究论文,该研究首次解析了拟南芥Dicer-like 1(DCL1)与底物RNA的复合物冷冻电镜结构,揭示了植物microRNA加工的分子机制,并为人类microRNA加工的机制研究提供了新的见解。MicroRNA是真核生物中广泛存在的一类非编码 RNA,它们通过结合靶基因mRNA抑制靶基因表达,从而参与真核生物基因表达调控。在动物中,由RNA聚合酶II转录产生microRNA初始转录产物pri-miRNA,其在细胞核内被Drosha切割产生microRNA前体pre-miRNA;运输到细胞质后,pre-miRNA被Dicer切割从而产生成熟的microRNA。在植物中,上述两步切割均由DCL1在细胞核内完成。DCL1具有与Dicer相似的结构域,那么它如何识别pri-miRNA和pre-miRNA并依次执行两步切割?为了回答这些问题,作者利用冷冻电镜单颗粒三维重构的方法,分别解析了拟南芥DCL1与pri-miRNA和pre-miRNA的复合物结构(图1)。
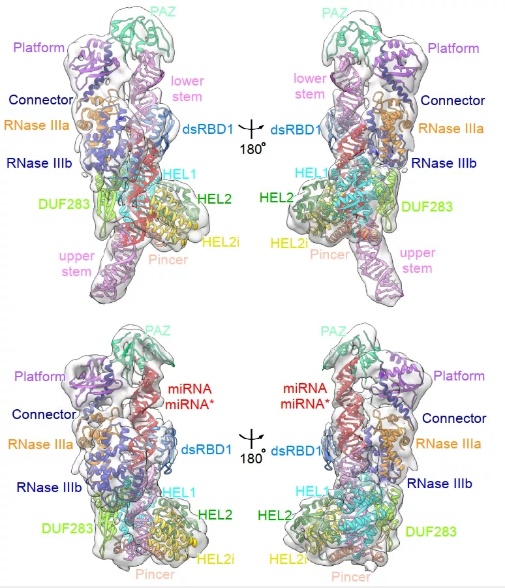
图1 DCL1-pri-miRNA(上)和DCL1-pre-miRNA(下)的结构
基于结构分析和生化实验验证,文章提出了植物microRNA加工的模型(图2)。首先,DCL1的PAZ结构域识别并结合pri-miRNA的单链区;同时,解旋酶结构域和DUF283结构域夹住pri-miRNA的双链区;然后,pri-miRNA的切割位点在dsRBD1结构域的协助下与RNase III活性中心对齐,执行第一步切割。完成第一步切割后,生成的pre-miRNA由ATP提供动力转位到PAZ结构域,再次将切割位点与RNase III活性中心对齐,从而执行第二步切割。
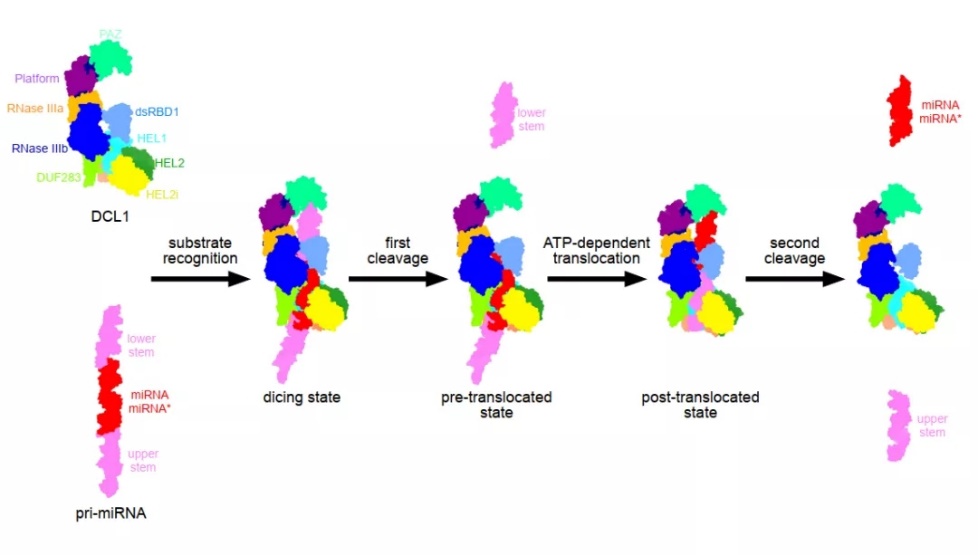
图2 植物microRNA加工的模型
2018年,Cell杂志报道了人类Dicer-pre-miRNA复合物的冷冻电镜结构(Liu et al., 2018)。出乎意料的是,结构中pre-miRNA远离RNase III活性中心,暗示其并非切割状态的结构。通过比较DCL1-pre-miRNA和Dicer-pre-miRNA的结构发现,DCL1的解旋酶结构域相对于Dicer的解旋酶结构域旋转了67°,因此作者推测Dicer的解旋酶结构域可能会发生巨大的构象变化并像DCL1一样夹住pre-miRNA从而完成切割。
浙江大学基础医学院博士后卫晓彬、博士研究生柯欢欢为该研究的共同第一作者,浙江大学基础医学院冯钰研究员为该研究的通讯作者。该研究的冷冻电镜工作全部在浙江大学冷冻电镜中心完成,也得到了浙江大学冷冻电镜中心高性能计算平台和医学院蛋白质平台支持。
 zju.edu.cn
zju.edu.cn